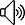主成分分析是一种常用的降维方法,多见于数据预处理阶段,其伪代码如下:
输入:样本数据集D={x1,x2,x3,x4},低空间维度数d'
过程:
-
1 对所有样本进行中心化(每个特征维度都减去其均值)
-
2 计算所有样本的协方差矩阵
-
3 对协方差矩阵进行特征分解
-
4 取最大的d'个特征值的对应特征向量w1,w2,w3
输出: 投影矩阵W*=(w1,w2,wd')
下面是其代码实现:
首先是产生数据,我们使用sklearn的make_blobs方法产生三类数据,每类有三个特征。数据结构是(n_samples, n_features)
图片如下:
可以看到,三维空间下的三类数据分的很开,在二维结构下,只有图(1)分的较开,从这一角度来看,我们的feature1和feature2是比较好的特征。
import numpy as np
from sklearn.datasets import make_blobs
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
np.random.seed(123)
if __name__ == '__main__':
# 产生三类数据
x, y = make_blobs(n_samples=600, n_features=3, centers=3)
# 绘制三维图像
fig = plt.figure(figsize=(8, 6))
ax = Axes3D(fig)
ax.scatter(x[:, 0], x[:, 1], x[:, 2], c='r')
plt.savefig('3-D distribution of Datapoints')
plt.show()
# # 绘制二维图像
fig = plt.figure(figsize=(8, 6))
ax1 = fig.add_subplot(2, 2, 1)
ax1.scatter(x[:, 0], x[:, 1], c=y)
ax2 = fig.add_subplot(2, 2, 2)
ax2.scatter(x[:, 1], x[:, 2], c=y)
ax3 = fig.add_subplot(2, 2, 3)
ax3.scatter(x[:, 0], x[:, 2], c=y)
plt.savefig('2-D distribution of Datapoints')
plt.show()
# step1 中心化
x_mean = np.mean(x, axis=0)
x_center = x - x_mean
# step2 计算协方差矩阵
covX = np.cov(x_center.T)
# step3 对协方差矩阵进行特征分解
featureValue, featureVector = np.linalg.eig(covX)
# step4 取最大的几个特征值
index = np.argsort(-featureValue)
selectVec = featureVector.T[index[:2]]
print(selectVec.shape)
final_matrix = np.dot(x_center, selectVec.T)
print(final_matrix.shape)
# 绘制最终二维图像
fig = plt.figure(figsize=(8, 6))
plt.scatter(final_matrix[:, 0], final_matrix[:, 1], c=y)
plt.title('2-D distribution after PCA')
plt.savefig('2-D distribution of Datapoints after PCA')
plt.show()
PCA的结果如下图所示,可以看到,效果明显:
总结分析:为什么PCA有效
分析:PCA的目标是通过某种线性投影方法,将高维的数据映射到较低维的空间中,并期望在所投影的维度上数据的信息量最大(方差最大,通过协方差矩阵的特征实现),具体内容可看博客:PCA主成分分析 详细解释